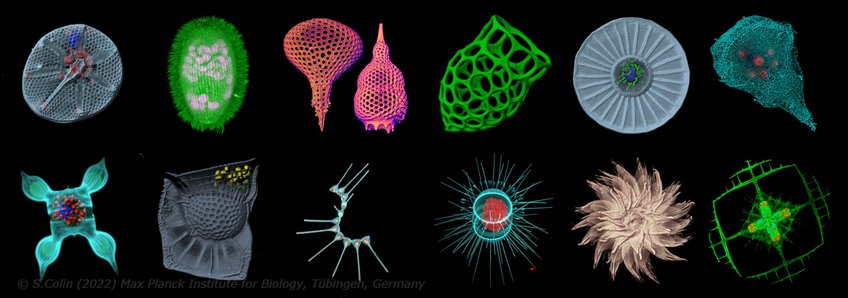
Cell Safari
Entwicklung eines phäno-genomischen Arbeitsablaufs zur Entschlüsselung der Biologie von Umweltprotisten unter Verwendung von 3D-Bildgebung in Kombination mit Omics-Technologien auf Einzelzellebene.
Protisten (einzellige Eukaryonten) repräsentieren die überwiegende Mehrheit der eukaryotischen Vielfalt, von denen nur wenige Linien die Vielzelligkeit entwickelt haben. Aufgrund der unglaublichen Bandbreite ihrer metabolischen Fähigkeiten, Morphologien, Lebenszyklen und Verhaltensweisen nehmen Protisten eine Schlüsselposition in globalen ökologischen Netzwerken ein sowohl als Primärproduzenten, Räuber, Mixotrophe, als auch als Symbionten und Parasiten.
Dennoch sind viele Arten nach wie vor nur unzureichend oder nur teilweise beschrieben, da sie hauptsächlich anhand von Umwelt-DNA nachgewiesen wurden. Infolgedessen fehlen immer noch wichtige biologische Schlüsselmerkmale (z. B. Morphologie, Lebenszyklus, biotische Interaktion). Unser Hauptziel ist es, die 3D-Mikroskopie mit Zellsortierverfahren zu kombinieren, um einen genau beschriebenen Phänotyp mit einem Genotyp auf Einzelzellebene in Verbindung zu bringen. Als Fallstudien konzentrieren wir uns auf Holobionten, die Protisten beinhalten, und noch spezifischer auf photosymbiotische Beziehungen.
- Viele entlegene Ökosysteme erfordern die Konservierung von Proben für Untersuchungen im Labor. Wir tragen zur Erforschung dreier Richtungen bei: (i) die Entwicklung von Live-Imaging-Tools für die Feldarbeit (z. B. In-Flow-Mikroskopie und Lebendzell-Färbung); (ii) die Optimierung der Probenkonservierung für korrelative Ansätze (z. B. Phänogenomik); und (iii) die Kultivierung von mikrobiellen Holobionten.
- Darüber hinaus bleibt die Korrelation zwischen fortschrittlicher Mikroskopie und genomischer Analyse eine Herausforderung für einzelne Zellen aus der Umwelt. Wir arbeiten an der Automatisierung eines bildbasierten Arbeitsablaufs zur Identifizierung und Sortierung von interessanten Objekten in hochkomplexen Partikelpopulationen. Unsere Herangehensweise verbessert die Genauigkeit der Zielerkennung und die Flexibilität des Sortierprozesses, allerdings auf Kosten eines geringeren Durchsatzes im Vergleich zu aktuellen Methoden. (z. B. FACS).
- Letztendlich glauben wir, dass phänogenomische Daten die Entwicklung von robusten Klassifizierungsmodellen für die KI-basierte Annotation von Hochdurchsatz-Mikroskopiedaten aus proben unterstützen werden.